

SCILAB à l'École nationale des ponts et chaussées
http://www.enpc.fr/scilab
Statistique
Analyse en composantes principales
Jean-François DELMAS et Saad SALAM
Date: dernière date de mise à jour : 19 novembre 2003
Considérons un nuage 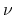 de
de 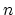 points dans un espace
points dans un espace 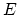 de dimension
de dimension 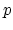 .
Lorsque
.
Lorsque 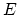 est de dimension élevée, on ne peut pas visualiser l'espace de
points. Le but de l'analyse en composantes principales est alors de
trouver le meilleur sous-espace
est de dimension élevée, on ne peut pas visualiser l'espace de
points. Le but de l'analyse en composantes principales est alors de
trouver le meilleur sous-espace 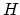 de
de 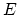 , de dimension
, de dimension 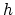 égale à 2 ou 3
par exemple, dans lequel on aura la meilleure représentation du nuage.
En fait, on va chercher à trouver le sous-espace
égale à 2 ou 3
par exemple, dans lequel on aura la meilleure représentation du nuage.
En fait, on va chercher à trouver le sous-espace 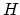 de dimension
de dimension 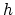 sur
lequel le nuage projeté du nuage
sur
lequel le nuage projeté du nuage 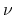 aura la plus grande "dispersion".
On cherchera donc à maximiser la somme des carrés des distances entre
tous les couples de points projetés.
aura la plus grande "dispersion".
On cherchera donc à maximiser la somme des carrés des distances entre
tous les couples de points projetés.
où 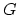 est le barycentre des points
est le barycentre des points 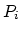 .
On voit donc que maximiser la somme des carrés des distances entre tous les couples de projetés des points équivaut à
maximiser la somme des carrés des distances entre les projetés des points et leur centre de gravité.
Notons
.
On voit donc que maximiser la somme des carrés des distances entre tous les couples de projetés des points équivaut à
maximiser la somme des carrés des distances entre les projetés des points et leur centre de gravité.
Notons
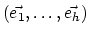 une base orthonormée de H.
une base orthonormée de H.
La matrice
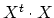 est symétrique réelle. C'est donc la matrice d'une forme quadratique Q dans une base
est symétrique réelle. C'est donc la matrice d'une forme quadratique Q dans une base 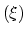 de E
et ainsi est diagonalisable dans une base orthonormée.
De plus, si on note
de E
et ainsi est diagonalisable dans une base orthonormée.
De plus, si on note
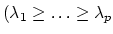 ) le spectre de
) le spectre de
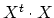 et
et
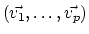 la base orthonormée de vecteurs propres associée, alors
la base orthonormée de vecteurs propres associée, alors
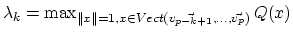 .
Alors, afin de maximiser l'expression précédente il faut choisir
.
Alors, afin de maximiser l'expression précédente il faut choisir
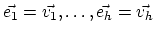 .
Dans ce cas,
.
Dans ce cas,
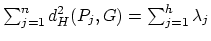 est l'inertie du nuage de points
est l'inertie du nuage de points 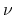 par rapport
au sous-espace H, et
par rapport
au sous-espace H, et
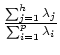 est le pourcentage d'inertie expliqué par
le sous-espace H. Ce pourcentage d'inertie rend compte de la part de dispersion du nuage
est le pourcentage d'inertie expliqué par
le sous-espace H. Ce pourcentage d'inertie rend compte de la part de dispersion du nuage  contenue dans le nuage
projeté de
contenue dans le nuage
projeté de  sur H.
Nous allons étudier dans cette partie la distribution des mesures de
poids de différentes parties d'un groupe de 23 bovins1 (cf la table ci-dessous).
Les variables représentent:
sur H.
Nous allons étudier dans cette partie la distribution des mesures de
poids de différentes parties d'un groupe de 23 bovins1 (cf la table ci-dessous).
Les variables représentent:
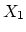 :
:- poids vif.
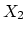 :
:- poids de la carcasse.
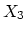 :
:- poids de la viande de première qualité.
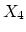 :
:- poids de la viande totale.
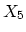 :
:- poids du gras.
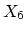 :
:- poids des os.
|
Bovin |
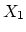 |
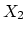 |
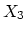 |
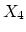 |
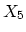 |
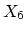 |
|
1 |
395 |
224 |
35.1 |
79.1 |
6.0 |
14.9 |
|
2 |
410 |
232 |
31.9 |
73.4 |
8.7 |
16.4 |
|
3 |
405 |
233 |
30.7 |
76.5 |
7.0 |
16.5 |
|
4 |
405 |
240 |
30.4 |
75.3 |
8.7 |
16.0 |
|
5 |
390 |
217 |
31.9 |
76.5 |
7.8 |
15.7 |
|
6 |
415 |
243 |
32.1 |
77.4 |
7.1 |
18.5 |
|
7 |
390 |
229 |
32.1 |
78.4 |
4.6 |
17.0 |
|
8 |
405 |
240 |
31.1 |
76.5 |
8.2 |
15.3 |
|
9 |
420 |
234 |
32.4 |
76.0 |
7.2 |
16.8 |
|
10 |
390 |
223 |
33.8 |
77.0 |
6.2 |
16.8 |
|
11 |
415 |
247 |
30.7 |
75.5 |
8.4 |
16.1 |
|
12 |
400 |
234 |
31.7 |
77.6 |
5.7 |
18.7 |
|
13 |
400 |
224 |
28.2 |
73.5 |
11.0 |
15.5 |
|
14 |
395 |
229 |
29.4 |
74.5 |
9.3 |
16.1 |
|
15 |
395 |
219 |
29.7 |
72.8 |
8.7 |
18.5 |
|
16 |
395 |
224 |
28.5 |
73.7 |
8.7 |
17.3 |
|
17 |
400 |
223 |
28.5 |
73.1 |
9.1 |
17.7 |
|
18 |
400 |
224 |
27.8 |
73.2 |
12.2 |
14.6 |
|
19 |
400 |
221 |
26.5 |
72.3 |
13.2 |
14.5 |
|
20 |
410 |
233 |
25.9 |
72.3 |
11.1 |
16.6 |
|
21 |
402 |
234 |
27.1 |
72.1 |
10.4 |
17.5 |
|
22 |
400 |
223 |
26.8 |
70.3 |
13.5 |
16.2 |
|
23 |
400 |
213 |
25.8 |
70.4 |
12.1 |
17.5 |
|
|
|
|
|
|
|
poids de taille  correspondant
aux poids des 23 bovins selon les 6 critères. (fichier
poids.txt). On
charge les données dans Scilab, après avoir sauvegardé localement le
fichier par exemple sous le nom
correspondant
aux poids des 23 bovins selon les 6 critères. (fichier
poids.txt). On
charge les données dans Scilab, après avoir sauvegardé localement le
fichier par exemple sous le nom poids.txt, à l'aide de la
commande
poids=fscanfMat("poids.txt").
L'analyse des données nous conduit tout d'abord à calculer les
paramètres descriptifs élémentaires presentés dans le tableau ci
dessous.
| |
Moyenne |
Écart-type |
Min |
Max |
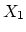 |
401.6 |
76.7 |
390.0 |
420.0 |
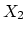 |
228.8 |
86.1 |
213.0 |
247.0 |
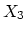 |
29.9 |
7.6 |
25.8 |
35.1 |
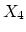 |
74.7 |
7.1 |
70.3 |
79.1 |
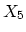 |
8.9 |
6.7 |
4.6 |
13.5 |
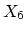 |
16.6 |
1.6 |
14.5 |
18.7 |
L'écart-type d'une suite de poids
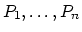 est estimé par:
est estimé par:
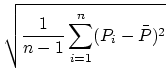 , où
, où
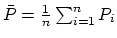 .
La matrice des corrélations nous donne une première idée des
associations existant entre les différentes variables.
.
La matrice des corrélations nous donne une première idée des
associations existant entre les différentes variables.
| |
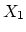 |
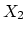 |
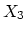 |
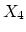 |
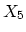 |
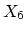 |
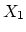 |
1.0000 |
0.6914 |
-0.0329 |
-0.0585 |
0.0820 |
0.0820 |
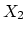 |
0.6914 |
1.0000 |
0.2837 |
0.3903 |
-0.3363 |
0.0917 |
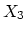 |
-0.0329 |
0.2837 |
1.0000 |
0.8948 |
-0.8773 |
0.0348 |
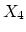 |
-0.0585 |
0.3903 |
0.8948 |
1.0000 |
-0.9016 |
0.0032 |
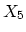 |
0.0820 |
-0.3363 |
-0.8773 |
-0.9016 |
1.0000 |
-0.3368 |
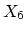 |
0.0820 |
0.0917 |
0.0348 |
0.0032 |
-0.3368 |
1.0000 |
La matrice de corrélations est obtenue en exécutant la fonction
correlation().
function c=correlation(A)
cov=covariance(A);
// vecteur contenant l'inverse des écarts-types
ectinv=(1)./sqrt(diag(cov));
// matrice des corrélations
c=diag(ectinv)*cov*diag(ectinv);
endfunction;
Cette fonction fait appel à la fonction covariance().
function cov=covariance(A)
// vecteur (1,p) des moyennes des colonnes (=variables)
moyenne = mean(A,"r");
// nombre de lignes dans la matrice A = nbre d'individus
n = size(A,"r")
// matrice recentrée
Acent = A - ones(n,1)*moyenne;
// matrice de covariance (Remarque: on a divisé par n-1 et non par n)
cov=(Acent'*Acent)/(n-1);
endfunction;
Calculons les valeurs propres de la matrice des corrélations et
intéressons nous aux pourcentages d'inertie.
| Axe |
Valeur propre |
Inertie |
Inertie cumule |
| 1 |
2.9914 |
49.90% |
49.90% |
| 2 |
1.6125 |
26.90% |
76.80% |
| 3 |
1.0387 |
17.30% |
94.10% |
| 4 |
0.2487 |
4.10% |
98.20% |
| 5 |
0.0758 |
1.30% |
99.50% |
| 6 |
0.0329 |
0.50% |
100.00% |
Figure 1:
Éboulis des valeurs propres
|
|
Les valeurs propres sont calculées avec la fonction val_prop().
function [c]=val_prop(A);
//renvoie les valeurs propres ordonnées de manière décroissante
[Diag,Base]=bdiag(A); // diagonalisation
// classement décroissant des valeurs propres
c=sort(diag(Diag));
endfunction;
L'inertie expliquée par la i-ème composante principale qui est associée
à la i-ème plus grande valeur propre est calculée avec la formule:
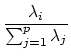 .
.
Question 1
Analyser le résultat obtenu.
|
Les vecteurs propres sont calculés avec la fonction vect_prop()
qui renvoie les vecteurs propres rangés dans l'ordre décroissant
des valeurs propres associées.
| |
1 |
2 |
3 |
4 |
5 |
6 |
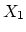 |
0.063 |
-0.743 |
0.060 |
0.597 |
0.283 |
-0.063 |
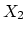 |
0.304 |
-0.609 |
0.117 |
-0.643 |
-0.331 |
0.019 |
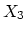 |
0.534 |
0.164 |
0.137 |
0.461 |
-0.646 |
0.200 |
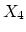 |
0.548 |
0.138 |
0.176 |
-0.130 |
0.595 |
0.528 |
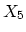 |
-0.552 |
-0.147 |
0.172 |
0.032 |
-0.193 |
0.778 |
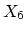 |
0.120 |
-0.100 |
-0.950 |
0.007 |
-0.040 |
0.266 |
function c=vect_prop(A);
// renvoie les vecteurs propres de A
// classés par valeurs propres corresp décroissantes
[Diag,Base]=bdiag(A);
// diagonalisation
if Base(:,1)==-abs(Base(:,1)) then Base=-1*Base; end;
// afin que les coef du 1er vect prop ne soient pas tous < 0.
[Valpr,k]=sort(diag(Diag));
// classement décroissant des valeurs propres
c = Base(:,k);
endfunction;
Nous obtenons les coordonnées des projetés des individus dans la base
orthonormée des vecteurs propres de la matrice des corrélations
avec la fonction acp_indiv().
function c=acp_indiv(A);
//Coordonnées des projetés des individus
// dans la base des vecteurs propres
mat_cor=correlation(A);
X=centrer_reduire(A);
VectP=vect_prop(mat_cor);
// les vecteurs propres
c = X*VectP;
// nouvelles coordonnees
endfunction;
Cette fonction fait appel à la fonction centrer_reduire() qui
permet de centrer et de normer une matrice de données de telle sorte que
la moyenne de chaque variable soit nulle et que son écart-type soit
égal à 1.
function c=centrer_reduire(A)
// permet de centrer et de réduire la matrice A
moyenne = mean(A,"r");
//vecteur (1,d) des moyennes des colonnes(=variables)
cov=covariance(A);
n = size(A,"r");
//nombre de lignes dans la matrice A=nbre d'individus
Acent = A - ones(n,1)*moyenne;
// A centre
c=Acent*(diag((1)./sqrt(diag(cov))));
endfunction;
Les coordonnées des points variables dans la base orthonormée des vecteurs propres sont obtenues avec
la fonction acp_var().
function c=acp_var(A);
//Coordonnées des variables dans le cercle des correlations
mat_cor=correlation(A);
X=centrer_reduire(A);
VectP=vect_prop(mat_cor);
// les vecteurs propres
ValP=val_prop(mat_cor);
// les valeurs propres
c = VectP*diag(sqrt(ValP));
endfunction;
Cela nous permet de représenter le nuage projeté du nuage initial de
poids et le cercle des corrélations dans le plan formé par deux
composantes principales quelconques.
Figure 2:
Projection des individus sur les axes principaux 1 et 2
|
|
Figure:
Cercle des corrélations pour les axes 1 et 2
|
|
Question 2
Que pouvez-vous dire sur le plan factoriel 1-2?
|
Pour représenter les coordonnées de 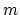 points de
points de
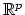 sur les axes
sur les axes
i-j, on
utilise la fonction nuage(A,i,j) où les lignes de la matrice
A sont les coordonnées des 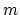 points de
points de
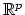 .
.
function nuage(Coord,i,j);
//projection des individus dans le plan i-j
xset("font",4,3);
deltax=(max(Coord(:,i))-min(Coord(:,i)))/20;
xmin=min(Coord(:,i))-deltax;
xmax=max(Coord(:,i))+deltax;
deltay=(max(Coord(:,j))-min(Coord(:,j)))/20;
ymin=min(Coord(:,j))-deltay;
ymax=max(Coord(:,j))+deltay;
// isoview(xmin,ymin,xmax,ymax);
titre="Projection sur le plan "+string(i)+"-"+string(j)
// afficher les individus
plot2d(Coord(:,i),Coord(:,j),-3,"031",rect=[xmin,ymin,xmax,ymax]);
xtitle(titre);
endfunction;
Pour représenter le cercle des corrélations sur les axes i-j, on
utilise la fonction cercle(A,i,j), où A est la matrice
des coordonnées des points variables dans la base orthonormée des
vecteurs propres.
function cercle(Coord,i,j)
xset("font",4,3);
Leg=['X1','X2','X3','X4','X5','X6'];
taille=6;
V=Coord(:,[i,j]);
V = V'.*.[1,0];
// insertion de l'origine
V = V';
p=size(V,"r");
//square(-1,-1,1,1);
t=[0:0.05:2*%pi]';
plot2d(cos(t),sin(t),1,"040");
xsegs([-1,1],[0,0]);
xsegs([0,0],[-1,1]);
titre="Cercle des corrélations sur le plan "+string(i)+"-"+string(j);
plot2d(V(:,1),V(:,2),5*ones(1,p),"000");
for k=1:taille, xstring(V(2*k-1,1),V(2*k-1,2),Leg(k));end;
endfunction;
Question 3
Que pouvez-vous dire sur le plan factoriel 2-3?
|
La qualité est représentée par le cosinus de l'angle entre le vecteur et
son projeté sur le plan factoriel considéré.
On utilise la fonction qualite_indiv(poids,i,j) pour
représenter la qualité des individus sur les plans factoriels
i-j.
function qualite_indiv(A,i,j);
//projection des individus dans le plan i-j
xset("font",4,3);
Coord=acp_indiv(A);
deltax=(max(Coord(:,i))-min(Coord(:,i)))/15;
xmin=min(Coord(:,i))-deltax;
xmax=max(Coord(:,i))+deltax;
deltay=(max(Coord(:,j))-min(Coord(:,j)))/15;
ymin=min(Coord(:,j))-deltay;
ymax=max(Coord(:,j))+deltay;
plotframe([xmin,ymin,xmax,ymax],[2,10,2,10],[%f,%f],..
["Projection sur le plan "+string(i)+"-"+string(j),"",""]);
X=centrer_reduire(A);
deff('[y]=qual_p(k)','y=( norm(Coord(k,[i,j])) / norm(Coord(k,:)))^2')
n=size(A,"r");
Q=feval(1:n,qual_p);
G=find(Q>0.9);
M=find(Q<0.9&Q>0.7);
B=find(Q<0.7);
for h=G,
xrects([Coord(h,i);Coord(h,j);0.2;0.2],3);
end;
for h=M,
xrects([Coord(h,i);Coord(h,j);0.1;0.1],2);
end;
for h=B,
xrects([Coord(h,i);Coord(h,j);0.05;0.05],1);
end;
xrects([xmin;ymin-0.7;0.2;0.2],3);
xstring(xmin+0.5,ymin-0.9,"cos^2>0.9")
xrects([xmin+2;ymin-0.7;0.1;0.1],2);
xstring(xmin+2.5,ymin-0.9,"0.7<cos^2<0.9")
xrects([xmin+5;ymin-0.8;0.05;0.05],1);
xstring(xmin+5.2,ymin-0.9,"cos^2<0.7")
endfunction;
Figure:
Qualité de la représentation des individus dans le plan
factoriel 1-2
|
|
Question 4
Quelle est votre analyse pour la qualité de la représentation sur les
plans 1-2 et 2-3?
|
Les données proviennent de deux races Charolais ou Zébu.
race=['C','C','C','C','C','C','C','C','C','C','C','C'];
race=[race,'Z','Z','Z','Z','Z','Z','Z','Z','Z','Z','Z'];
Le programme barycentres(acp_indiv(poids),C,i,j) permet de
visualiser la variable nominale supplémentaire race dans le plan
factoriel i-j.
function barycentres(Coord,C,i,j);
// preparation du graphique
xbasc();
deltax=(max(Coord(:,i))-min(Coord(:,i)))/20;
xmin=min(Coord(:,i))-deltax;
xmax=max(Coord(:,i))+deltax;
deltay=(max(Coord(:,j))-min(Coord(:,j)))/20;
ymin=min(Coord(:,j))-deltay;
ymax=max(Coord(:,j))+deltay;
plotframe([xmin,ymin,xmax,ymax],[2,10,2,10],[%f,%f],..
["Projection sur le plan "+string(i)+"-"+string(j),"",""]);
plot2d([0,0],[ymin,ymax],2,"000");
plot2d([xmin,xmax],[0,0],2,"000");
// modalités
mod=['C','Z'];
// representation des individus suivant leur modalite
for k=mod,
I=find(C==k);
B=mean(Coord(I,:),"r");
B=B'.*.[1,0];
B=B'
plot2d(B(:,i),B(:,j),5,"000");
plot2d(B(:,i),B(:,j),-1,"000");
xset("font",2,3);
xstring(B(1,i),B(1,j),k);
xset("font",2,1);
for l=I,
xstring(Coord(l,i),Coord(l,j),k);
end;
end;
endfunction;
Question 5
Analyser le rôle de la variable nominale.
|
Figure:
Variable supplémentaire dans le plan factoriel 1-2
|
|
Notes
- ... bovins1
- source
INRA



Jean-Francois Delmas
2003-11-19
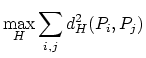
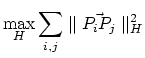
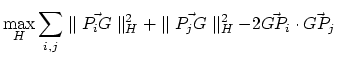
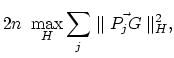
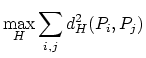
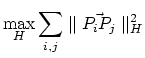
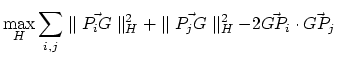
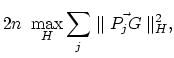
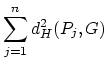
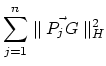
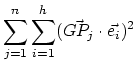
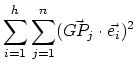
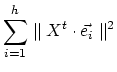
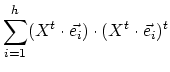
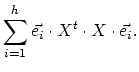
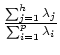 est le pourcentage d'inertie expliqué par
le sous-espace H. Ce pourcentage d'inertie rend compte de la part de dispersion du nuage
est le pourcentage d'inertie expliqué par
le sous-espace H. Ce pourcentage d'inertie rend compte de la part de dispersion du nuage 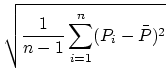 , où
, où
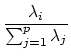 .
.